- RNAseq RNA-seq 기반 유전자 발현 프로파일링
-
- Date 2019-01-30
RNA-seq 기반 유전자 발현 프로파일링
RNAseq 데이터를 이용해 원하는 대사 경로의 특정 유전자 발현 값의 패턴 분석
RNAseq 분석을 통해 표준유전체가 완료된 생물뿐만 아니라 진행 중인 생물체를 대상으로 관심 있는 현상에 관여하는 유전자가 무엇인지, 유전자 발현 변화와 패턴을 분석합니다.
시료 준비 과정이나 시퀀싱 과정 중에 발생되는 오차를 최대한 보정하고, 염기서열의 특징에 의해 발생되는 오류 가능성을 최소화하기 위한 다양한 통계 기법을 적용하여 정확도를 높이기 위한 노력을 기울이고 있습니다. 단순 유전자 발현값 측정에 머무르지 않고 목표 형질이나 현상에 관련된 유전자를 선발하기 위해 clustering 기법, GO, KEGG 등 다양한 기법과 문헌자료를 통해 구축한 씨더스 고유 DB를 활용하여 보다 정제된 데이터를 드리고자 노력하고 있습니다.
RNAseq Gene expression profiling
1. DEG & Clustering 분석
◆ DEG (Differentially Expressed Genes) 선발
◆ DEG (Differentially Expressed Genes) 선발
샘플별 유전자 발현값을 통계적으로 처리하여 대조군과 비교군 샘플 간에 발현이 유의한 DEG 후보군을 선발합니다.
<유전자 발현값을 표현한 MA Plot>
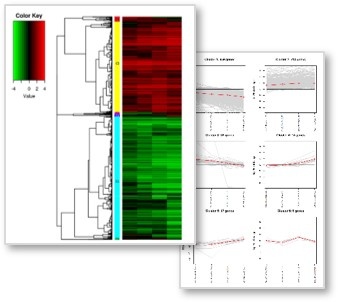
◆ Clustering 분석
탐색한 DEG 후보군의 발현 패턴에 따라 군집화(Clustering) 하여 그래프로 나타냅니다.
탐색한 DEG 후보군의 발현 패턴에 따라 군집화(Clustering) 하여 그래프로 나타냅니다.
2. GO & KEGG pathway 분석
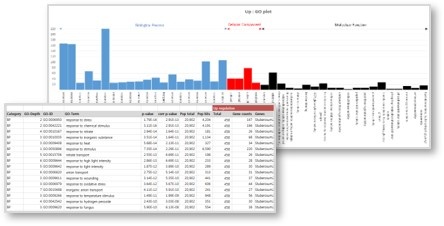
◆ GO (Gene Ontology) 분석 선발
선발된 DEG는 씨더스가 보유한 Gene Ontology DB와 대조하여 Gene annotation을 수행합니다.
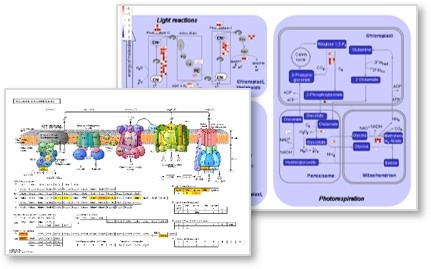
◆ KEGG pathway 분석
KEGG mapper, Mapman 등의 visualization tool을 이용해 목표 Pathway에 관여하는 유전자 발현의 변화를 한 눈에 확인할 수 있습니다.
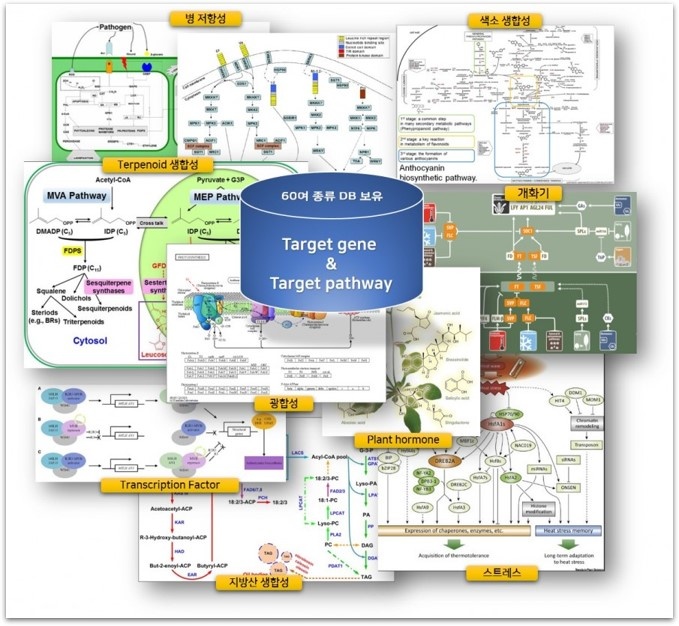
병저항성 관련, 스트레스 관련, TF 관련, 식물 호르몬 관련, 지방산 생합성 관련, 개화 관련, Terpenoid 생합성 관련, 과실색 생합성 관련, 광합성 관련 유전자군 등 다양한 기능별 유전자군을 문헌자료 통해 정리 후 씨더스 고유 DB로 구축하여 RNAseq 분석에 활용합니다.
Reference.
• Chaudhary R, Atamian HS (2017) Resistance-Gene-Mediated Defense Responses against Biotic Stresses in the Crop Model Plant Tomato. J Plant Pathol Microbiol 8:4
• Reinprecht Y, Yadegari Z, Perry GE, Siddiqua M, WrightLC, McClean PE and Pauls KP (2013) In silico comparison of genomic regions containing genes coding for enzymes and transcription factors for the phenyl-propanoid pathway in Phaseolus vulgaris L. and Glycine max L.Merr. Front.PlantSci. 4:317.
• Yan Liu, Shi-Hong Luo, Axel Schmidt, Guo-Dong Wang, Gui-Ling Sun, Marcus Grant, Ce Kuang, Min-JieYang, Shu-Xi Jing, Chun-Huan Li, Bernd Schneider, Jonathan Gershenzon, Sheng-Hong Li The Plant Cell Mar 2016, 28 (3) 804-822
• https://www.genome.jp/kegg-bin/show_pathway?map=map00195&show_description=show
• https://www.creative-proteomics.com/services/plant-hormone-analysis-service.htm
• http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-54052010000400002
• http://www.phytosystems.ulg.ac.be/florid/
• Liu Y, Tikunov Y, Schouten RE, Marcelis LFM, Visser RGF and Bovy A (2018) Anthocyanin Biosynthesis and Degradation Mechanisms in Solanaceous Vegetables
• Liu Y, Huang Z, Ao Y, Li W, Zhang Z (2013) Transcriptome Analysis of Yellow Horn (Xanthoceras sorbifolia Bunge): A Potential Oil-Rich Seed Tree for Biodiesel in China. PLOS ONE 8(9):
• Ohama N, Sato H, Shinozaki K, Yamaguchi-Shinozaki K.(2017) Transcriptional Regulatory Network of Plant Heat Stress Response. Trends Plant Sci. 2017 Jan;22(1):53-65.
• Chaudhary R, Atamian HS (2017) Resistance-Gene-Mediated Defense Responses against Biotic Stresses in the Crop Model Plant Tomato. J Plant Pathol Microbiol 8:4
• Reinprecht Y, Yadegari Z, Perry GE, Siddiqua M, WrightLC, McClean PE and Pauls KP (2013) In silico comparison of genomic regions containing genes coding for enzymes and transcription factors for the phenyl-propanoid pathway in Phaseolus vulgaris L. and Glycine max L.Merr. Front.PlantSci. 4:317.
• Yan Liu, Shi-Hong Luo, Axel Schmidt, Guo-Dong Wang, Gui-Ling Sun, Marcus Grant, Ce Kuang, Min-JieYang, Shu-Xi Jing, Chun-Huan Li, Bernd Schneider, Jonathan Gershenzon, Sheng-Hong Li The Plant Cell Mar 2016, 28 (3) 804-822
• https://www.genome.jp/kegg-bin/show_pathway?map=map00195&show_description=show
• https://www.creative-proteomics.com/services/plant-hormone-analysis-service.htm
• http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-54052010000400002
• http://www.phytosystems.ulg.ac.be/florid/
• Liu Y, Tikunov Y, Schouten RE, Marcelis LFM, Visser RGF and Bovy A (2018) Anthocyanin Biosynthesis and Degradation Mechanisms in Solanaceous Vegetables
• Liu Y, Huang Z, Ao Y, Li W, Zhang Z (2013) Transcriptome Analysis of Yellow Horn (Xanthoceras sorbifolia Bunge): A Potential Oil-Rich Seed Tree for Biodiesel in China. PLOS ONE 8(9):
• Ohama N, Sato H, Shinozaki K, Yamaguchi-Shinozaki K.(2017) Transcriptional Regulatory Network of Plant Heat Stress Response. Trends Plant Sci. 2017 Jan;22(1):53-65.


