- Genome 돌연변이 계통의 Structural variations 분석
-
- Date 2018-12-20
돌연변이 계통의 Structural variations 분석
구조변이 (Structural variation)는 일반적으로 동일한 종의 개체들 사이에서 결실(deletion), 삽입(insertion), 중복(duplication), 역위(inversion) 및 전좌(translocation)를 포함한 변이를 말합니다. 단일 염기 다형성 (single nucleotide polymorphism; SNP)이나 small insertion/deletion 변이 보다 개체 간 유전 변이의 가장 큰 원인으로 인식되고 있으며, SV는 유전적 다양성 및 표현형 변이와 질병에 주요한 원인이 될 수 있다고 보고되었습니다.
이러한 SV 연구의 중요성이 증가함에 따라 씨더스에서는 다년간의 생물정보 분석 경험과 노하우를 통해 structural variation 분석 결과를 제공합니다.
▶ 왜 NGS를 이용하여 분석하는가?
차세대 시퀀싱 (NGS) 기술은 DNA 서열 변이를 포괄적으로 분석할 수 있는 기회를 제공합니다. SNP, small In/Del뿐만 아니라 50bp이상의 구조 변이(Structural variation; SV) 분석에도 사용됩니다.
NGS 데이터는 SV 탐지를 위한 새로운 접근 방법으로, array comparative genome hybridization (aCGH) 방식에 비해 다양한 유형의 변이와 크기 탐지를 가능하게 합니다. 또한 breakpoints 식별을 가능하게 하여 돌연변이 메커니즘을 유추하는 등 후속 분석에 활용 될 수 있습니다.
▶ 언제 SV를 분석 하는가?
인위적으로 방사선을 조사한 돌연변이 계통에서 특히 SV 분석을 많이 활용하고 있습니다. 돌연변이 육종(Mutation Breeding)은 중요한 작물 육종 기술의 하나로서, 우수한 품종에 방사선을 처리하여 특정 형질만을 변화시켜 신품종 육성의 소재로 이용하는 등 최근에 많이 사용하는 방법입니다.
감마선(gamma-rays), 양성자 빔(proton beam), 이온화 방사선 (ionizing radiation) 등 다양한 유형의 방사선 처리에 따라, 또는 방사선 선량 강도에 따라 발생하는 돌연변이의 유형과 크기가 다양하기 때문에 SNP, small InDel과 함께 염색체의 구조적 이상을 탐지할 수 있는 SV분석을 많이 연구하는 추세입니다.
▶ 어떻게 SV를 탐색하는가?
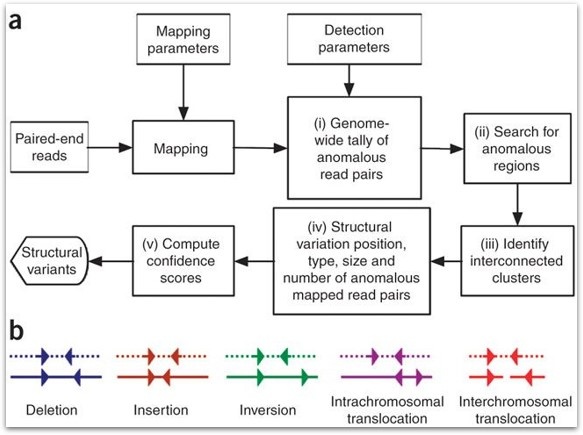
씨더스에서는 다양한 분석 툴 중에서 일반적으로 read-pair 방식을 이용하는 BreakDancer (Chen et al., 2009)를 사용하여 분석하고 있습니다.
BreakDancer는 비정상적인 read의 방향성을 갖거나 insert size의 크기를 갖는 anomalously mapped read pairs (ARPs)를 식별하여 총 5가지 유형 (insertions, deletions, inversions, intra- and inter-chromosomal translocations)의 SV를 예측할 수 있습니다.
▶ 씨더스만의 노하우?
SV 탐색 툴을 사용하면 무수히 많은 false-positive SVs 결과를 얻게 됩니다. 분석 기술이 발달했음에도 불구하고 신뢰성 있는 SV의 검출은 여전히 어려운 과제입니다. 따라서 ARP read count와 confidence score 등의 필터를 거쳐 정제된 SV를 확보하는 것이 SV 분석의 중요 포인트 입니다. 또한 BreakDancer 이외에 다른 분석 툴을 함께 사용하여 SV를 정제하는 방법도 사용하고 있습니다. 이러한 다년간의 생물정보 분석 경험과 고민을 통해 얻은 노하우를 적용하여 정제된 SV결과를 제공합니다.
▶ 어떤 결과를 주는가?
- 원품종 대비 돌연변이 계통에서 확보한 SV결과에서 정제된 후보를 제공합니다.
- VCF (variant call format) 형식으로 결과를 제공합니다.
- SV정확도 판별을 위해 supporting read-pair 개수와 서열을 제공합니다.
- Breakpoints를 통해 변이 위치 식별 및 크기를 확인할 수 있고, 주변 서열을 함께 제공합니다.
- Breakpoints 주변 유전자를 확인하여 gene mutations 여부 및 해당 유전자의 gene ontology (GO) 분석을 통한 유전자 기능 분석을 제공가능합니다.
- SV 결과를 가시화할 수 있도록 mapping bam 파일을 함께 제공합니다.

IGV (Integrated Genome Viewer; Broad Institute)를 이용한 Deletion structural variant 가시화 예시 (Noll AC, et al. 2016)
▶ Referecne
- Chen K, et al. (2009) BreakDancer: an algorithm for high-resolution mapping of genomic structural variation. Nat. Methods.
- Noll AC, et al. (2016) Clinical detection of deletion structural variants in whole-genome sequences. Npj Genomic Medicine.
- Koboldt DC, et al. (2012) Massively parallel sequencing approaches for characterization of structural variation. Methods Mol Biol.


